Род Orbivirus семейства Reoviridae насчитывает 22 вируса, а также 7 еще не классифицированных изолятов, являющихся кандидатами на признание их самостоятельными видами на основании серологических и филогенетических характеристик [1–9].
С экологической точки зрения все орбивирусы являются арбовирусами и способны инфицировать широкий круг позвоночных хозяев: диких и домашних животных, летучих мышей, птиц, а некоторые – также и человека. В качестве переносчиков и хозяев орбивирусов могут выступать комары, москиты, мокрецы и клещи [2, 10–13]. Традиционно наибольшее внимание исследователей уделялось 4 представителям рода Orbivirus, вызывающим экономически значимые заболевания сельскохозяйственных животных: вирусам синего языка овец (Bluetongue virus, BTV), эпизоотической геморрагической лихорадки (Epizootic hemorrhagic disease virus, EHDV), африканской чумы лошадей (African horse sickness, AHSV) и энцефалита лошадей (Equine encephalosis virus, EEV) [14]. В то же время роль орбивирусов в патологии человека до сих пор слабо изучена. Для части орбивирусов, передающихся клещами, показана способность вызывать лихорадочные состояния и неврологические нарушения при инфицировании человека [14, 15]. Однако их вклад в структуру заболеваемости инфекциями, передающимися клещами, остается неизвестным. Одним из таких патогенов является вирус Кемерово (KEMV), для которого доказана способность вызывать энцефалит у человека [15, 16].
Вирус Кемерово был выделен в 1962 г. группой советских и чехословацких вирусологов под руководством академика М.П. Чумакова из ликвора пациента с энцефалитом, возникшим после присасывания клеща, а также из клещей вида Ixodes persulcatus [15].
Позже были открыты другие близкородственные вирусу Кемерово виды, рассматриваемые в настоящее время как группа вирусов Грейт-Айленд [17].
До недавнего времени считалось, что ареалом вируса Кемерово является территория Западной Сибири [18]. Однако в исследованиях, проведенных с применением молекулярных методов, показано, что территория циркуляции вируса Кемерово значительно шире и включает не только Сибирь, но и Урал, а также отдельные территории Европейской части Российской Федерации [19]. При этом вирус Кемерово обнаруживается, в том числе, в клещах вида Ixodes ricinus, являющихся переносчиками близкородственного вируса Трибеч (Tribec virus, TRBV), циркуляция которого выявлена на территории Восточной Европы, а также в Молдавии и на юге Украины [20, 21].
Изучение генетической вариабельности вируса Кемерово и других представителей группы Грейт-Айленд затруднено ввиду малого количества расшифрованных полногеномных последовательностей.
Целью нашей работы стала расшифровка геномов штаммов вируса Кемерово, изолированных на территории Российской Федерации в разные годы из разных источников, и сравнение их с ранее опубликованными последовательностями штаммов вируса Кемерово и других представителей группы вирусов Грейт-Айленд.
Материалы и методы
Культивирование вируса
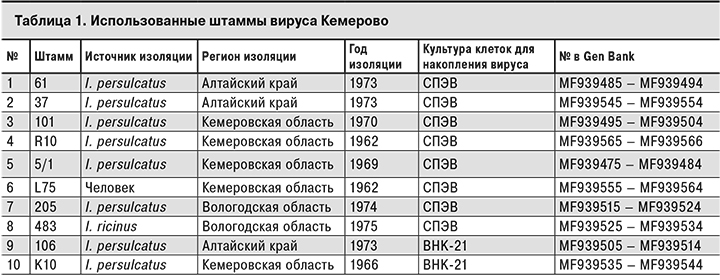
В работе были использованы 10 штаммов вируса Кемерово из коллекции ФГБНУ ««Федеральный научный центр исследований и разработки иммунобиологических препаратов им. М.П. Чумакова» РАН, полученные в разное время из разных источников, в том числе от человека (табл. 1).
Вирус размножали в перевиваемой культуре клеток почки эмбриона свиньи (СПЭВ) или в культуре клеток почки сирийского хомячка BHK-21. Вирус наращивали в зараженной культуре в течение 48–72 ч при 37 ˚С, после чего содержащую вирус культуральную жидкость отбирали для последующего выделения РНК.
Выделение РНК
Вируссодержащую культуральную жидкость освобождали от клеточного дебриса центрифугированием при 10 000 об/мин в течение 30 мин при 4 ˚С. Супернатант центрифугировали при 25 000 об/мин в течение 4 ч при 4 ˚С. Супернатант удаляли, осадок вирусных частиц тщательно ресуспендировали в 600 мкл Tрис-Натрий-ЭДТА-буфера [10 мМ Трис, 100 мМ хлорида натрия, 1 мМ этилендиаминтетраацетата (ЭДТА); pH = 8,0] и добавляли 10% (вес/объем) додецилсульфата натрия до конечной концентрации 1%.
РНК дважды экстрагировали смесью фенола и хлороформа (в объемном соотношении 1:1), затем отделяли водную фазу и добавляли 5 M ацетат аммония до конечной концентрации 1 M и 3 объема 96% этанола. Полученную смесь тщательно встряхивали, выдерживали в течение 1 ч при -80 оС, после чего РНК осаждали центрифугированием. Осадок отмывали 80% этанолом, высушивали в эксикаторе под вакуумом и растворяли в стерильной воде.
Качество полученной РНК оценивали электрофоретическим методом в 1% агарозном геле с добавлением бромистого этидия.
Для избавления от балластной ДНК 9 мкл исходной РНК обрабатывали 2 Ед ДНКазы I («Fermentas», Латвия) с добавлением 1,2 мкл 10Х реакционного буфера («Fermentas», Латия) при 37 ˚С в течение 30 мин. Инактивацию фермента осуществляли добавлением 25 мM ЭДТА («Fermentas», Латия) с последующей инкубацией смеси 10 мин при 65 оС. Полученный продукт очищали с помощью набора QIAmp MinElute PCR Purification Kit («Qiagen» Германия) по протоколу производителя. Очищенную РНК элюировали в 10 мкл свободной от РНКаз воды RNAse-free water («Qiagen», Германия).
Вирусную РНК извлекали методом преципитации в 2 M хлориде лития. К 5 мкл очищенной РНК после обработки ДНКазой I добавляли 20 мкл 8 M хлорида лития, доводили объем до 80 мкл свободной от РНКаз воды RNAse-free water и инкубировали на льду в течение 16 ч. Затем центрифугировали 30 мин при 13 000 об/мин. Для последующей работы отбирали супернатант, содержащий дцРНК, который очищали с помощью набора QIAmp MinElute PCR Purification Kit по протоколу производителя. Очищенную РНК элюировали в 10 мкл свободной от РНКаз воды RNAse-free water.
Получение библиотек вирусной кДНК
Для получения библиотек вирусной кДНК реализовали подход, описанный S. Maan и соавт. [22], с некоторыми модификациями, схематично представленный на рис. 1.
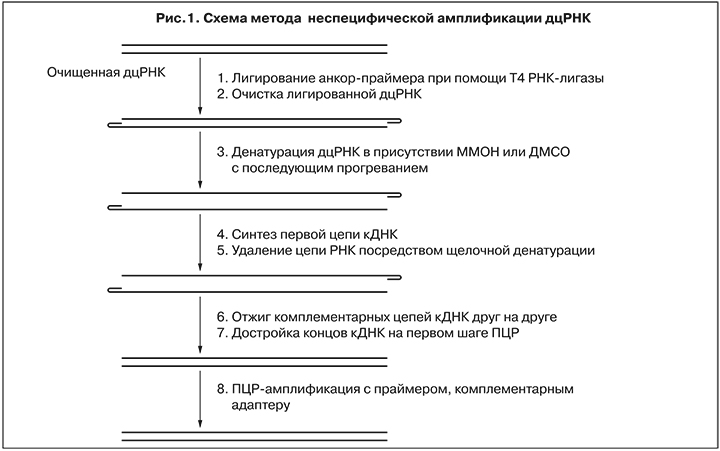
На первом этапе производили лигирование петлевых адаптеров Reo-Sp-Loop – «анкор-праймеров» с помощью Т4 РНК-лигазы.
Реакционная смесь объемом 20 мкл содержала 10 мкл очищенной дцРНК, 25 пмоль адаптера Reo-Sp-Loop (5’p-GAC CTC TGA GGA TTC TAA AC_Sp9_T CCA GTT TAG AAT CC-3’), 2 мкл 10Х реакционного буфера для Т4 РНК-лигазы («Fermentas», Латвия), 4 мкл 50% полиэтиленгликоля (PEG4000; «Fermentas», Латия), 10 Ед. Т4-РНК-лигазы («Fermentas», Латвия). До необходимого объема смесь доводили свободной от РНКаз водой RNAse-free water.
Смесь инкубировали при 25 ˚С в термошейкере при 400 об/мин. Затем продукт лигирования очищали с помощью набора QIAmp MinElute PCR Purification Kit по протоколу производителя, РНК элюировали в 10 мкл свободной от РНКаз воды RNAse-free water.
5 мкл дцРНК с лигированными адаптерами при в присутствии 3 мкл ДМСО («Fermentas», Латия) денатурировали под слоем минерального масла при 90 ˚С в течение 2 мин. Затем пробы извлекали из термостата и быстро помещали в лед. На льду под масло вносили по 12 мкл реакционной смеси, состоящей из 4 мкл 5Х буфера для AMV-ревертазы («Fermentas», Латия), 1 мкл 10mM dNTP mix («Thermo Fisher Scientific Inc.», США), 6 мкл воды RNAse-free water. Реакцию проводили в термошейкере при 37 ˚С в течение 30 мин, перемешивая смесь при 580 об/мин. Далее добавляли еще по 20 Ед AMV-ревертазы («Fermentas», Латия) и продолжали инкубировать еще 30 мин при 42 оС. Продукт обратной транскрипции очищали с помощью набора QIAmp MinElute PCR Purification Kit по протоколу производителя. Полученную первую цепь кДНК элюировали в 10 мкл свободной от РНКаз воды RNAse-free water.
Достройку второй цепи кДНК и амплификацию полученных фрагментов проводили в одношаговом режиме с праймером pReo, комплементарным адаптеру (5’p-GTT TAG AAT CCT CAG AGG TC-3’). Реакционная смесь общим объемом 25 мкл содержала 2 мкл кДНК, 10 пмоль праймера pReo, 2,5 мкл дНТФ 1,76 мМ (ФБУН «Центральный НИИ эпидемиологии» Роспотребнадзора, далее – ФБУН ЦНИИЭ, Россия), 10 мкл ПЦР-смесь-2 blue (ФБУН ЦНИИЭ, Россия). До нужного объема смесь доводили стерильной деионизированной водой (ФБУН ЦНИИЭ, Россия). Реакцию осуществляли с помощью ДНК-амплификатора «Терцик» («ДНК-технология», Россия) в следующем режиме: 95 ˚С – 3 мин; 95 ˚С – 20 с, 72 ˚С – 15 мин (n = 2); далее 95 ◦С – 20 с, 50 ˚С – 20 с, 72 ˚С – 1 мин (n = 45); 72 ˚С – 3 мин.
Полученный ПЦР-продукт очищали с помощью набора QIAmp MinElute PCR Purification Kit по протоколу производителя с элюцией в 15 мкл. Качество библиотек оценивали методом электрофореза в 1,5% агарозном геле с добавлением бромистого этидия.
Подготовку библиотек для высокопроизводительного секвенирования проводили с помощью набора реагентов Nextera XT DNA Library Preparation Kit («Illumina Inc.», США). Качество полученных библиотек оценивали, используя 2100 Electrophoresis Bioanalyser («Agilent Tecnologies Inc.», США). Cеквенирование осуществляли с помощью платформы для высокопроизводительного секвенирования MiSeq («Illumina Inc.», США).
In silico анализ
Данные, полученные по результатам произведенного секвенирования, картировали на референсный геном, в качестве которого была взята полногеномная последовательность штамма вируса Кемерово 21/10 (Gen Bank KC288130-KC288139).
Выравнивание нуклеотидных последовательностей производили с помощью пакета программ MEGA версии 5.2 с применением алгоритма MUSCLE [23]. С помощью этого же пакета программ осуществляли филогенетический анализ. Филогенетические деревья строили методом максимального правдоподобия (maximum likelihood estimation) с использованием параметрической модели GTR (General Time-Reversible model) и оценкой гамма-распределения вариации частот между сайтами и долей инвариантных сайтов в последовательности [24, 25]. При проведении филогенетического анализа были использованы последовательности геномов других представителей рода Orbivirus.
Идентичность нуклеотидных последовательностей всех сегментов генома для штаммов вируса Кемерово, а также сегмента 2 для представителей группы Грейт-Айленд вычисляли, используя программное обеспечение CLC Genomics Workbench версии 3.6.5.
Результаты
Секвенированы и аннотированы полные геномы 9 штаммов вируса Кемерово. Для штамма KEMV R10 секвенированы и охарактеризованы сегменты, кодирующие последовательность белков VP1 (полимераза) и VP3. Все полученные нуклеотидные последовательности депонированы в базе данных Gen Bank (см. табл. 1).
Длины сегментов генома совпадают у всех изученных штаммов, за исключением сегмента 5, кодирующего неструктурный белок NS1. В некодирующей части на 5’-конце данного сегмента для штамма KEMV 61 обнаружена вставка в 1 пару нуклеотидов (п. н.), а для штамма KEMV k10 – делеция в 1 п. н. Таким образом, длина сегмента 5 для указанных штаммов составляет 1720 и 1718 п. н. соответственно.
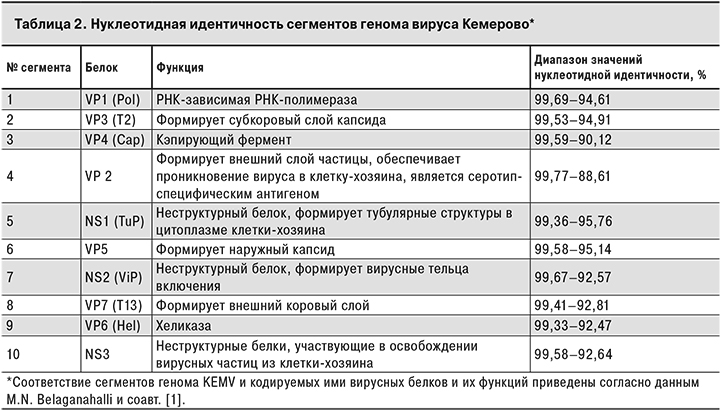
Вычислен процент нуклеотидной идентичности для всех сегментов генома штаммов вируса Кемерово (табл. 2).
Для сегментов 1 и 2 генома изучаемых штаммов и близкородственных представителей группы Грейт-Айленд выполнен филогенетический анализ (рис. 2).

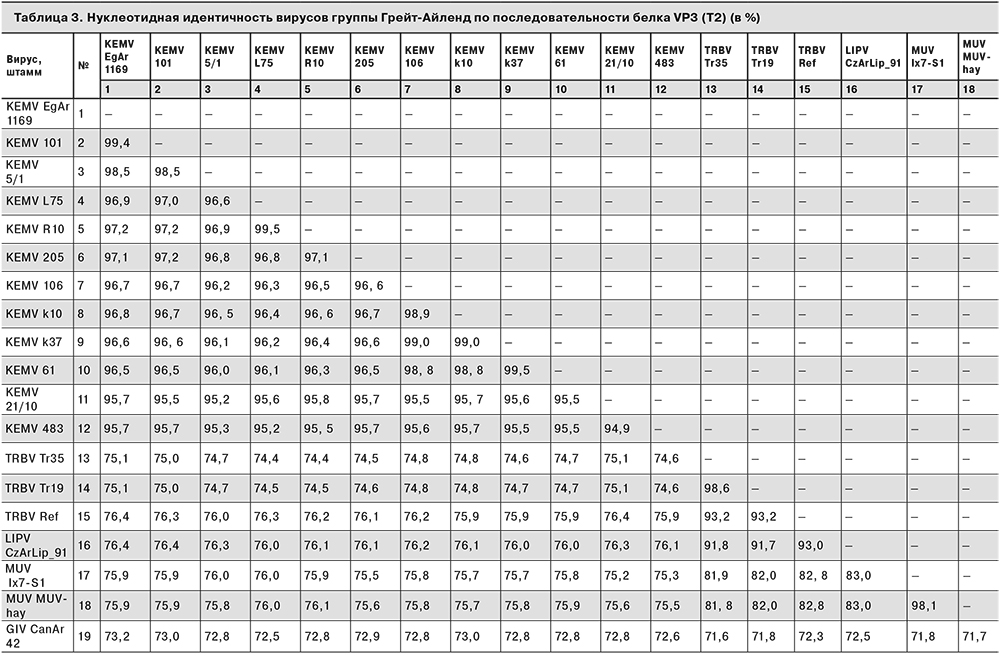
По результатам сравнения идентичности нуклеотидных последовательностей штаммов вируса Кемерово и близкородственных представителей группы Грейт-Айленд по сегменту генома, кодирующему белок VP3 (T2), все штаммы вируса Кемерово продемонстрировали oт 94,9 до 99,5% сходства друг с другом и от 95,7 до 99,3% сходства по отношению к референсному штамму EgAr-1169. Штаммы вируса Трибеч показывают от 93,2 до 98,6% нуклеотидной идентичности друг с другом, от 74,3 до 76,4% – со штаммами вируса Кемерово и от 91,6 до 93,1% – с вирусом Липовник. Нуклеотидная идентичность вирусов Муко и Трибеч по указанному сегменту составляет от 81, 8 до 83,1% (табл. 3).
Обсуждение
Данные анализа основных молекулярно-генетических характеристик полученных геномов, таких как длина сегментов, (G + C)-состав, длина и структура концевых фрагментов, а также процент нуклеотидной идентичности (см. табл. 2) указывают на несомненную принадлежность всех исследуемых штаммов к одному виду.
Согласно полученным данным, самым вариабельным сегментом генома вируса Кемерово является сегмент 4, кодирующий белок наружного слоя капсида VP2 [1]. Этот белок является антигеном, определяющим серотип вируса, а также отвечает за вирулентность. Самыми консервативными являются сегменты, кодирующие неструктурный белок NS1 и структурные белки капсида VP5 и VP3(T2) [1].
Все штаммы, кроме KEMV 106 и KEMV k10, имеют альтернативную рамку считывания в сегменте 9. Альтернативные рамки считывания в сегменте 9, кодирующем фермент хеликазу, описаны и у других представителей группы Грейт-Айленд, однако их функция на сегодняшний день окончательно не выяснена. Существуют предположения о влиянии альтернативной рамки считывания в сегменте 9 на патогенность вируса для млекопитающих [14].
Филогенетический анализ полученных нуклеотидных последовательностей по первым двум сегментам генома подтвердил, что все исследуемые штаммы вируса Кемерово формируют отдельную ветвь в пределах группы Грейт-Айленд, отличную от ветви, формируемой штаммами вируса Трибеч, в которую, помимо собственно вируса Трибеч, входят также вирус Липовник и вирус Муко, впервые изолированный и описанный в 2015 г. на территории Японии [26].
Штаммы KEMV демонстрируют тесную взаимосвязь. Наиболее интересной оказывается филогенетическая позиция штамма EgAr-1169, выделенного из аргасового клеща на территории Египта в 1961 г. Филогенетическая реконструкция положения данного штамма на филогенетическом древе внутри клады KEMV указывает на возможную связь со штаммами KEMV 101 и 205, полученными из клещей I. persulcatus на территории Кемеровской и Вологодской областей соответственно в 70-х годах прошлого века, что может свидетельствовать об общности их происхождения. Это позволяет предположить, что штамм EgAr-1169 мог быть занес ен на территорию Египта из Западной Сибири в ходе миграции мелких воробьинообразных, поскольку пути их перелета проходят по этим территориям [27, 28].
KEMV L75, единственный сохранившийся штамм, выделенный в Кемеровской области от больного человека с симптомами энцефалита в 1962 г., оказывается филогенетически наиболее близок штамму R10, полученному в тот же временной период из клеща I.persulcatus на территории Кемеровской области.
В соответствии с положениями IX Доклада Международного комитета по таксономии вирусов, существует ряд критериев принадлежности представителей рода Orbivirus к одному виду. Одним из них является идентичность нуклеотидной последовательности сегмента, кодирующего ген субкорового белка T2 (VP3): более 76% идентичности для представителей одного вида, менее 74% – для представителей разных видов [17]. Согласно полученным значениям нуклеотидной идентичности по сегменту 2, кодирующему последовательность белка VP3, вирусы Кемерово и Трибеч укладываются в заданные критерии принадлежности к одному виду. В пользу предположения, что вирусы Кемерово и Трибеч являются не самостоятельными видами, а разными геновариантами одного вида, также говорят ранее полученные данные о антигенном сходстве KEMV и TRBV [20, 29, 30] и наличии общего переносчика [19]. Очевидно, что для окончательных выводов о таксономической принадлежности вирусов Кемерово и Трибеч необходимо проанализировать большее число штаммов, а также охарактеризовать возможность их симпатрической циркуляции и формирования реассортантных форм.
Результаты сравнения нуклеотидной идентичности вирусов группы Грейт-Айленд, входящих в кладу TRBV, позволяют предположить, что все они в действительности также могут представлять собой один полиморфный вид, широко распространенный на территории Евразии – от Восточной Европы (TRBV), через территорию России и Дальний Восток (KEMV), до островов Японии (MUV). Столь широкая географическая распространенность вируса может быть обеспечена за счет передачи посредством птиц как непосредственных переносчиков клещей и промежуточных хозяев вируса. Это положение согласуется с данными, имеющимися для других орбивирусов, в том числе для некоторых представителей группы Грейт-Айленд [14], однако требует более тщательного рассмотрения.
Выводы
По результатам расшифровки последовательностей геномов 10 штаммов вируса Кемерово существенно дополнена информация о генетическом разнообразии вируса. Тем самым сформирована основа для дальнейших исследований вируса Кемерово как потенциального объекта в рамках изучения проблемы новых и возвращающихся инфекций.
Для более детального понимания сложных взаимоотношений вирусов внутри группы необходимы современные штаммы KEMV и TRBV, а также подробные эпидемиологические исследования циркуляции вирусов в природе.



