Streptococcus (S.) pneumoniae и Haemophilus (H.) influenzae могут как бессимптомно колонизировать слизистые верхних дыхательных путей (ВДП), так и являться причиной серьезных инфекционных заболеваний и осложнений гриппа и ОРВИ у детей, лиц пожилого возраста, беременных, пациентов с хроническими заболеваниями, а также у иммунокомпрометированных пациентов. В «доковидную эру» пневмококк вызывал основную массу случаев внебольничной пневмонии (ВП) [1]. В зависимости от возраста, вакцинального статуса, а также возможных факторов риска S. pneumoniae может являться причиной от 5 до 88% случаев ВП у лиц разного возраста [2–4]. В этиологической структуре ВП определенную роль также играет H. influenzae (10–20% случаев) [5, 6]. Как S. pneumoniae, так и H. influenzae обнаруживают у 70–80% больных острым средним отитом (ОСО) [7, 8]. Кроме этого, пневмококк и гемофильная палочка могут вызывать бактериальные менингиты [9, 10].
Носоглоточное носительство S. pneumoniae и H. influenzae затрудняет этиологическую диагностику инфекционных заболеваний. В разгар сезона ОРВИ пневмококк обнаруживают более чем у 70% здоровых людей, у детей доля носителей может достигать 97% [11]. Гемофильная палочка колонизирует ВДП 20–80% здоровых детей [12, 13].
В последние годы противоэпидемические меры, принимаемые в первый год пандемии COVID-19, способствовали снижению инвазивных инфекций, вызванных S. pneumoniae и H. influenzae [14]. Несмотря на это частота встречаемости пневмококка у здоровых детей младше 5 лет снизилась лишь незначительно [15]. Вместе с тем показано, что колонизация пневмококком у пациентов с COVID-19 ослабляет иммунную защиту организма [16].
В лабораторной практике S. pneumoniae и H. influenzae идентифицируют стандартными бактериологическими методами, чувствительность которых может снижаться в связи с нарушением условий хранения и транспортировки биоматериала, а также проведением антибиотикотерапии. Диагностику также затрудняют особенности культуральных свойств пневмококка и гемофильной палочки. Например, в связи с наличием мутаций некоторые штаммы пневмококка проявляют нехарактерные для них биохимические свойства и, соответственно, могут быть ошибочно идентифицированы как стрептококки группы Viridans [17]. H. haemolyticus часто определяют как нетипируемые штаммы H. influenzae (NTHi). H. haemolyticus редко является причиной ВП, ОСО и менингита, тогда как NTHi могут вызывать эти инфекции, особенно в странах, где посредством вакцинации удалось снизить циркуляцию гемофильной палочки типа b [18]. Показано, например, что пневмококк в крови выявляют менее чем в 5% случаев ВП у детей, а в плевральной жидкости – менее чем в 20% случаев [19]. По данным государственного статистического наблюдения в РФ, пневмококковая этиология ВП подтверждается лишь в 1,3% случаев [20].
Несмотря на повсеместное применение MALDI-ToF (Matrix-Assisted Lazer Desorption/Ionization Time-of-Flight – матрично-активированная лазерная десорбция/ионизация с время-пролетным разделением) масс-спектрометрии, обеспечивающей быструю идентификацию возбудителя, до сих пор остаются трудности в дифференциации пневмококка и его близкородственных видов. Для повышения специфичности метода в качестве дополнительных прибегают к биохимическим исследованиям [21, 22]. При дифференциации H. influenzae и H. haemolyticus методом MALDI-ToF масс-спектрометрии могут возникнуть сложности в связи со схожестью их белковых профилей [23]. Существенным недостатком этого метода является необходимость получения культуры микроорганизма.
Полимеразная цепная реакция в режиме реального времени (ПЦР-РВ) – это наиболее быстрый, удобный и доступный метод идентификации микроорганизмов. Показано, что ПЦР-РВ является высокочувствительным и специфичным методом в отношении идентификации ДНК S. pneumoniae и H. influenzae в образцах мокроты [24], плазмы крови [25], бронхо-альвеолярного лаважа (БАЛ) [26] и плевральной жидкости [27]. Кроме этого, возможность разработки количественного формата ПЦР-РВ позволяет оценить концентрацию возбудителя в биологическом материале, тем самым выявляя случаи носительства.
Цель исследования – разработка набора реагентов для количественного определения ДНК S. pneumoniae и H. influenzae методом ПЦР-РВ и валидация его на биологическом материале пациентов с ВП среднего и тяжелого течения, а также сравнение результатов ПЦР и других методов лабораторной диагностики.
Материалы и методы
Были протестированы несеротипированные изоляты и штаммы (тип b) H. influenzae (101) и изоляты S. pneumoniae (181) серотипов 2, 3, 4, 6A, 6B, 7FA, 8, 9NL, 9V, 9VA, 10А, 11А, 11AD, 14, 15AF, 15В, 15BC, 18, 18ABCF, 19A, 19F, 22, 22FA, 23B, 23F, 37. Аналитическую специфичность набора доказывали при исследовании ДНК близкородственных видов: S. pyogenes, S. agalactiae, S. oralis, S. salivarius, S. mitis, S. anginosus, H. parainfluenzae, H. parahaemolyticus.
В работе исследовали биологический материал 1140 больных [мазки со слизистой оболочки носо- и ротоглотки, мокрота, БАЛ, жидкость из полости среднего уха, спинномозговая жидкость (ликвор), цельная венозная кровь, тканевой (аутопсийный) материал], полученный в ходе работы Референс-центра по мониторингу за инфекциями верхних и нижних дыхательных путей (НДП) ФБУН «Центральный НИИ эпидеимиологии» Роспотребнадзора (далее – ЦНИИЭ). В исследование проспективно включали 60 взрослых пациентов с верифицированным диагнозом ВП тяжелого течения, не получавших антимикробной терапии. У пациентов собирали респираторные образцы (60 образцов; мокрота или трахеальный аспират/БАЛ) для бактериоскопии, культурального исследования и исследования методом ПЦР; образец мочи (58 образцов) для проведения быстрых тестов с целью выявления растворимых антигенов L. pneumophila серогруппы 1 и S. pneumoniae). Культивирование и идентификацию микроорганизмов проводили с помощью стандартных методов и процедур [28].
Исследовали биологические образцы (мокрота/аспират из трахеи) 66 детей с рентгенологически подтвержденной ВП средней тяжести. Дополнительно протестированы парные образцы биологического материала из ВДП и НДП 44 пациентов.
Экстракцию нуклеиновых кислот (НК) проводили с использованием комплекта реагентов «РИБО-преп» (ЦНИИЭ). Ген bexA в изолятах Haemophilus выявляли набором реагентов для ПЦР-РВ «АмплиСенс® N. meningitidis/H. influenzae/S. pneumoniae-FL» (ЦНИИЭ). Для установления этиологии ВП дополнительно использовали наборы реагентов для ПЦР-РВ: «АмплиСенс® MRSA-скрин-титр-FL», «АмплиСенс®Streptococcus pyogenes-скрин/монитор-FL», «АмплиСенс® Mycoplasma pneumoniae/Chlamydophila pneumoniae-FL», «АмплиСенс® Legionella pneumophila-FL», «АмплиСенс® ОРВИ-скрин-FL», «АмплиСенс® Influenza virus A/B-FL», «АмплиСенс® Influenza virus A-тип-FL», «АмплиСенс® Influenza virus А/H1-swine-FL» того же производителя.
Культивирование и идентификацию микроорганизмов проводили в соответствии со стандартными методами и процедурами.
Фрагменты ДНК генов 16S рРНК, rpoB и hpd амплифицировали методом ПЦР с визуализацией результатов в агарозном геле. Определение нуклеотидной последовательности ПЦР-продуктов (табл. 1) проводили набором ABI PRISM Big Dye™ v.3.1 (Applied Biosystems, США). Сборку и анализ последовательностей осуществляли с использованием пакета программ Lasergene v.10.1 (DNASTAR Inc., США).
Результаты
В качестве мишени для S. pneumoniae был выбран локус Spn9802, для H. influenzae – ген hpd. Аналитическую чувствительность и линейный диапазон определяли тестированием повторов разведений плазмид, содержащих вставку фрагментов геномов S. pneumoniae и H. influenzae, в образцах биологического материала. Аналитическая чувствительность составила 103 копий/мл, линейный диапазон – от 104 до 108 копий/мл.
При тестировании образцов ДНК близкородственных микроорганизмов, перечисленных выше, и ДНК человека неспецифических реакций выявлено не было.
Для всех культур, определенных в бактериологическом исследовании как S. pneumoniae, были получены положительные результаты ПЦР-РВ.
Для 4 изолятов H. influenzae (№ 125, 213, 806 и 1739) получен отрицательный результат ПЦР-РВ. Результаты ПЦР с набором реагентов «АмплиСенс® N. meningitidis/H. influenzae/S. pneumoniae-FL», где в качестве мишени выступал ген bexA, также были отрицательны для этих изолятов. ПЦР-продукты, полученные в результате амплификации фрагментов генов 16S рРНК, rpoB и hpd исследуемых изолятов, были секвенированы. Изоляты № 125 и 1739 по результатам секвенирования генов 16S рРНК, rpoB и hpd были отнесены к H. haemolyticus. По результатам секвенирования фрагмента rpoB изоляты № 213 и 806 принадлежат к нетипируемым штаммам H. influenzae, однако мы не получили фрагмента гена hpd нужной длины в ПЦР (табл. 2). При этом были амплифицированы фрагменты разной длины, секвенирование которых показало высокую (до 93%) гомологию различным участкам генома H. influenzae. Таким образом, мы сделали заключение, что тестируемые изоляты № 213 и 806 – это H. influenzae с делетированным геном hpd.
При исследовании мокроты от 60 пациентов с ВП культура S. pneumoniae была выделена из 14 (23%) образцов, а методом ПЦР-РВ ДНК S. pneumoniae обнаружили в 38 (63%) образцах. Положительные результаты бактериологического исследования и ПЦР-РВ совпали в 100% случаев. Дополнительно методом ПЦР-РВ удалось установить пневмококковую этиологию для 24 пациентов. У всех 16 больных с положительным результатом экспресс-теста на наличие антигена пневмококка в моче ДНК S. pneumoniae была обнаружена в ПЦР-исследовании и в мокроте. Дополнительно у 22 больных с отрицательным результатом экспресс-теста на наличие антигена пневмококка в моче методом ПЦР-РВ была определена ДНК S. pneumoniae в мокроте.
Методом ПЦР-РВ ДНК H. influenzae обнаружили у 19 (32%) больных ВП. При этом культура была выделена только из одного образца мокроты, содержащего ДНК H. influenzae в высокой концентрации (108 копий/мл).
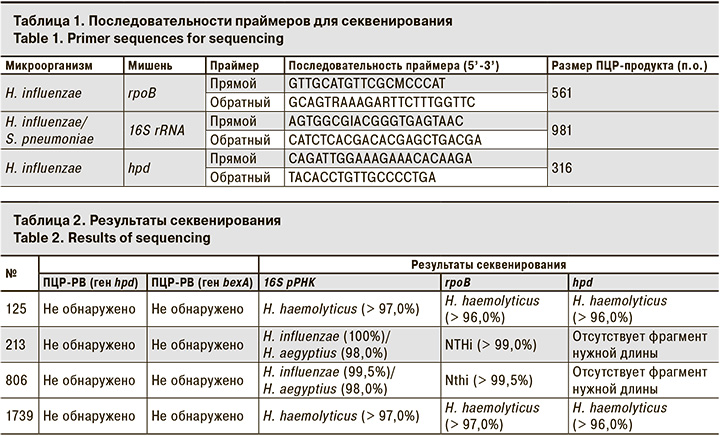
Этиологию ВП средней тяжести у 66 детей удалось установить в 91% случаев (рис. 1).
Наибольший вклад в развитие ВП внесли M. pneumoniae (37,9%) и респираторно-синцитиальный вирус (Human Orthopneumovirus, ранее носивший название Human Respiratory Syncytial virus, RSV – 6,7%). Треть всех случаев составили микст-инфекции, где S. pneumoniae и H. influenzae наиболее часто обнаруживали в ассоциациях с вирусами или другими бактериями.
S. pneumoniae были выявлены у 11 (17%) детей, концентрация ДНК составила от 104 до 3 x 108 копий/мл.
H. influenzae были обнаружены в 15 (23%) случаях в диапазоне концентраций ДНК 105–109 копий/мл.
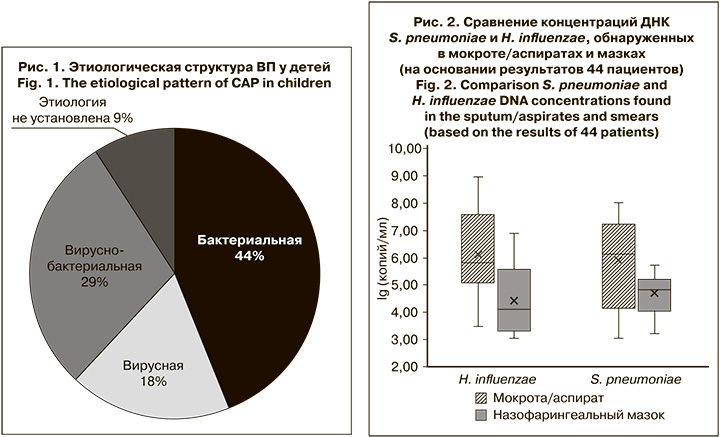
При исследовании парных образцов биологического материала от 44 детей с ВП ДНК S. pneumoniae и H. influenzae выявляли в мокроте/аспиратах чаще и в более высокой концентрации по сравнению с респираторными мазками (медиана: 106 и 7 x 105 копий/мл vs 7x104 и 104 копий/мл соответственно) (рис. 2).
У 4 и 7 пациентов соответственно ДНК S. pneumoniae и H. influenzae не были обнаружены в назофарингеальных мазках. При этом диапазон концентраций ДНК S. pneumoniae в образцах мокроты этих пациентов составил 5 x 103–107 копий/мл, H. influenzae – 5 x 103–109 копий/мл.
У одного пациента при наличии ДНК S. pneumoniae в респираторном мазке в концентрации 104 копий/ мл в образце из НДП этот возбудитель обнаружен не был. При этом в образце мокроты выявили РНК респираторно-синцитиального вируса и ДНК H. influenzae в концентрации 106 копий/ мл. У другого в образце из ВДП была выявлена ДНК H. influenzae в концентрации 3 x 105 копий/мл, а в мокроте не обнаружены НК ни одного из искомых вирусных или бактериальных возбудителей. Таким образом, эти случаи можно расценивать как случаи транзиторного носительства.
Обсуждение
В данной работе представлены результаты разработки набора реагентов в формате ПЦР-РВ для количественного определения ДНК S. pneumoniae и H. influenzae и валидация его на биологических образцах пациентов с ВП.
К настоящему времени на основе ПЦР существуют различные методики для выявления ДНК S. pneumoniae и H. influenzae. Наиболее распространенные мишени для идентификации пневмококка методом ПЦР – гены lytA, ply и cpsA. Однако первые 2 в связи с присутствием в близкородственных видах снижают специфичность метода [29], а ген капсида (cpsA) отсутствует в нетипируемых S. pneumoniae (non-typeable strains, NTPn). Так как последовательности 16S рРНК S. pneumoniae и близкородственных видов Streptococcus spp. имеют высокую гомологию (≈100%) [30], могут наблюдаться ложноположительные реакции. Для локуса Spn9802 показана высокая положительная предсказательная ценность при установлении диагноза пневмококковой пневмонии [25]. Однако следует отметить, что S. pseudopneumoniae также содержит локус Spn9802 [30]. Поскольку S. pseudopneumoniae идентифицируют достаточно редко [30], невозможность отличить его от пневмококка не является существенным ограничением для использования локуса Spn9802, который и был нами выбран в качестве мишени для ПЦР-РВ.
В связи с обменом генетической информацией между представителями рода Haemophilus [31] бывает достаточно сложно выбрать мишень для ПЦР, которая бы позволила надежно дифференцировать близкородственные виды. Так, существуют методики для определения ДНК H. influenzae, где областью амплификации являются фрагменты генов bexA, hpd, iga, ompP2, ompP6, fucK и 16S рРНК. Гены 16S рРНК у разных видов Haemophilus обладают высоким полиморфизмом [32]. Кроме этого, типирование по гену 16S рРНК предполагает этап секвенирования [33]. Бескапсульные штаммы невозможно идентифицировать, если в качестве мишени в ПЦР использовать ген bexA [34]. Гены ompP2 и ompP6 показали низкую чувствительность и специфичность в ПЦР [33, 35]. При детекции гена fucK возможны ложноположительные реакции, если в биологическом образце присутствуют H. parahaemolyticus, H. parainfluenzae или P. multocida. А ген iga не позволяет отличать H. influenzae от H. parainfluenzae и N. meningitidis [36].
Для нашего набора в качестве мишени для H. influenzae был выбран ген hpd в связи с возможностью дифференцировать H. influenzae и H. haemolyticus [23], а также наличием этого гена в большинстве патогенных нетипируемых штаммов. Показано, что протеин D, кодируемый геном hpd, является одним из ключевых факторов патогенности бескапсульных штаммов H. influenzae [37, 38]. Необходимо отметить, что только 6% NTHi не содержат ген hpd [39]. Среди исследованных нами изолятов 2% штаммов не имели ген hpd.
В нашей выборке бактериологическим методом два изолята H. haemolyticus определены как H. influenzae. Это можно объяснить тем, что H. haemolyticus могут терять способность к гемолизу в процессе культивирования [23], в связи с чем до 40% их идентифицируют как NTHi [40].
В ПЦР-РВ в образцах больных тяжелой ВП пневмококк выявляли в 2 и 3 раза чаще, чем экспресс-тестом на наличие антигена S. pneumoniae в моче и бактериологическим методом соответственно, что говорит о более высокой диагностической эффективности ПЦР.
В структуре возбудителей ВП тяжелого течения преобладал S. pneumoniae (63%), H. influenzae обнаруживали у 32% пациентов.
Культура S. pneumoniae была выделена только из образцов мокроты пациентов, содержащих высокую концентрацию ДНК возбудителя (медиана – 3 x 107 копий/ мл). У пациентов, в образцах мочи которых обнаруживали антиген, концентрация ДНК S. pneumoniae в мокроте также была достаточно высокой (медиана – 8 x 106 копий/мл). А при низкой нагрузке (медиана – 5 x 104 копий/мл) ДНК S. pneumoniae в мокроте культура уже не выделялась, как и не обнаруживался антиген в моче у этих больных, что говорит о меньшей чувствительности этих методов по сравнению с ПЦР.
В своем исследовании мы показали более высокую информативность биологического материала из НДП по сравнению с материалом из ВДП при установлении этиологии ВП средней тяжести у детей. С целью дифференциации пневмококковой этиологии ВП от носительства исследователи во главе с K. Stralin [41] сравнивали результаты ПЦР для крови и мокроты с результатами бактериологического исследования этого биологического материала, а также экспресс-теста на антиген пневмококка в моче. Ими был сделан вывод, что наибольшая чувствительность и специфичность обеспечиваются при пороговом значении концентрации ДНК S. pneumoniae 106 копий/мл в мокроте, при этом для назофарингеальных аспиратов трудно было определить порог значимости результатов.
Заключение
В результате применения набора реагентов для количественного определения ДНК S. pneumoniae и H. influenzae методом ПЦР-РВ с наборами реагентов для выявления основных вирусных и бактериальных возбудителей ОРИ и гриппа этиология пневмонии была установлена у 91% детей. Вирусная этиология ВП составила 18%, вирусно-бактериальная – 29% и бактериальная – 44%. Показано, что наиболее информативным для диагностики является материал из НДП. Используя ПЦР-РВ, диагностику пневмококковой пневмонии тяжелого течения у взрослых удалось улучшить в 2–3 раза. S. pneumoniae преобладал в структуре таких пневмоний.
Разработанный набор реагентов АмплиСенс® Пневмо-квант-FL (РУ № РЗН 2022/16467), адаптированный для различных видов биологического материала (мазки со слизистой оболочки носо- и ротоглотки, мокрота, БАЛ, жидкость из полости среднего уха, ликвор, цельная кровь, аутопсийный материал), может быть рекомендован для этиологической диагностики инфекций, вызванных S. pneumoniae и H. influenzae.
Необходимо продолжать исследования по определению порога значимой концентрации ДНК условно-патогенных бактерий для дифференциации заболевания от носительства.



