Human betaherpesvirus 6A и 6B (ВГЧ-6A, ВГЧ-6B) распространены среди людей во всем мире [1].
Все больше данных свидетельствуют о том, что ВГЧ-6A и ВГЧ-6B могут интегрировать в субтеломерную/теломерную область хромосомы клетки хозяина и наследоваться через зародышевую линию. Такая форма существования вирусов становится возможной благодаря особенностям строения их геномов [2, 3], а именно строению 2 терминальных областей (Т1, Т2), фланкирующих уникальную область (U). Т1 и Т2 содержат совершенные и несовершенные повторы, комплементарные теломерным повторам хромосом человека. Это позволяет вирусам встраивать свой геном посредством гомологичной рекомбинации. При интеграции в половые клетки возможна дальнейшая передача хромосомно-интегрированного (хи) вируса по наследству от одного или обоих родителей с вероятностью 50% [2, 4, 5]. В таком случае наследуемый хиВГЧ-6А/В является эндогенным и выявляется во всех клетках человека-носителя, обладающего хиВГЧ-6А/В-статусом, в соотношении не менее 1:1 (ядросодержащая клетка человека : геном вируса).
На современном этапе основным критерием лабораторного подтверждения активной ВГЧ-6А/В-инфекции, требующей назначения противовирусной терапии, является обнаружение высокой концентрации ДНК вируса в крови пациентов с помощью методов амплификации нуклеиновых кислот. Однако при лабораторном обследовании лиц с хиВГЧ-6А/В-статусом такой подход может приводить к диагностическим ошибкам, необоснованному назначению и излишнему применению лекарственных препаратов в рамках проведения фармакотерапии [6, 7].
Согласно последним опубликованным зарубежным данным [8], от 0,6 до 2% населения, в зависимости от географического положения региона, в котором выполнялась выборка, являются носителями наследуемого хиВГЧ-6А/В. В Российской Федерации подобного рода исследования по изучению распространенности наследуемого хиВГЧ-6А/В ранее не проводились, что и явилось целью настоящей работы.
Материалы и методы
В период с мая 2017 по июнь 2018 г. проведено лабораторное обследование 262 клинически здоровых лиц, проживающих в Москве и Московской области. Среди них были 75 мужчин и 187 женщин в возрасте от 18 до 67 лет (медиана возраста составила 36 лет). Кроме того, для расшифровки случая наследственной передачи хиВГЧ-6В в исследование были дополнительно включены 3 члена одной семьи: двое мужчин и женщина в возрасте 21 года, 50 и 69 лет соответственно Лабораторное обследование проводили в соответствии с законодательством РФ, международными этическими нормами, нормативными документами ФБУН «Центральный НИИ эпидемиологии» Роспотребнадзора (далее – ЦНИИЭ) при наличии информированного добровольного письменного согласия пациентов.
Материалом для лабораторного исследования служили образцы биологического материала: кровь, мазки из ротоглотки, моча, волосяные фолликулы, ногтевые пластины. При проведении скрининга, направленного на изучение распространенности хиВГЧ-6А/В, тестировали образцы крови. При установлении факта наследственной передачи эндогенного вируса кроме цельной крови дополнительно исследовали и другие виды биологического материала, перечисленные ранее.
Экстракцию ДНК из анализируемых образцов биологического материала проводили при помощи комплекта реагентов «РИБО-преп» (РУ № ФСР 2008/03147, ЦНИИЭ, Россия). Количественное определение ДНК ВГЧ-6А/В выполняли методом ПЦР в режиме реального времени (ПЦР-РВ) с использованием набора реагентов «АмплиСенс®HHV6-скрин-титр-FL» (РУ № ФСР 2010/09506, ЦНИИЭ, Россия). Постановку и анализ результатов амплификации проводили на приборе с системой детекции флуоресцентного сигнала в режиме реального времени Rotor-Gene Q (Qiagen, Германия) в соответствии с инструкцией производителя. Концентрацию ДНК ВГЧ-6А/В рассчитывали в логарифмах копий ДНК вируса на стандартное количество (105) клеток человека, оцененное по β-глобиновому гену.
Полногеномное секвенирование образцов тотальной ДНК выделенных российских клинических изолятов хиВГЧ-6В, выполняли после предварительного обогащения вирусного генома с использованием панели праймеров, предложенной E. Zhang с соавт. [9]. Условия проведения амплификации и составы реакционных смесей описаны ранее [10]. По окончании амплификации смесь ампликонов чистили с помощью магнитных карбоксилированных частиц и изготовляли библиотеки для секвенирования. Секвенирование выполняли на платформе Miseq (Illumina, США), используя наборы MiSeq Reagent Kit v2 или v3 (Illumina, США). Недостающие фрагменты генома хиВГЧ-6В амплифицировали отдельно с последующим секвенированием по Сэнгеру. Полученные прочтения фильтровали по качеству и удаляли последовательности специфических праймеров и адаптеров. Достаточность объема полученных данных оценивали путем картирования прочтений на референсный геном эталонного экзогенного штамма Human betaherpesvirus 6В HST (acc. AB021506, длина 161573 нуклеотидов), депонированного в GenBank NCBI, с последующим анализом глубины секвенирования и определением процента перекрытия. При достаточном объеме анализируемых данных проводили de novo сборку контигов с помощью программы Spades v3.0.15. Псевдогеном получали путем картирования собранных контигов на референсный геном с помощью программы Blast и объединения ориентированных контигов поли-N-линкерами в одну последовательность.
Для сравнительного филогенетического анализа использовали 28 последовательностей штаммов/изолятов ВГЧ-6B (GenBank NCBI), выделенных ранее в разных странах мира: Великобритании, Вьетнаме, Италии, Кении, Республике Заир, Китае, Пакистане, Перу, США, Финляндии, Японии. Среди них было 2 эталонных экзогенных и 26 эндогенных геномов штаммов/изолятов. Филогенетическую реконструкцию выполняли путем картирования прочтений или последовательностей геномов (полученных в результате проведенного секвенирования и депонированных ранее в GenBank NCBI) на референсный геном и выявлением однонуклеотидных вариаций. Вариации, локализованные в областях рекомбинации и областях, содержащих повторы, удаляли из анализа. Филогенетическое дерево для исследуемых образцов строили на основе полученной матрицы нуклеотидных вариаций с помощью программного обеспечения RAxML с использованием модели нуклеотидных замен с гамма-распределением по вариабельным позициям (GTR + Γ). Достоверность узлов оценивали с помощью метода статистического бутстрэп-анализа с количеством повторов 1000.
 Статистический анализ полученных результатов включал обработку данных с использованием компьютерной программы «Microsoft Excel 2016».
Статистический анализ полученных результатов включал обработку данных с использованием компьютерной программы «Microsoft Excel 2016».
Результаты
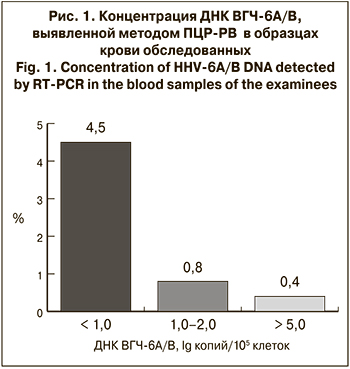
В ходе работы ДНК ВГЧ-6A/B в крови обнаружена у 15 из 262 обследованных (5,7% случаев). У 247 (94,3%) человек ДНК вируса в крови не обнаружена. При этом определяемая концентрация ДНК вируса в крови обследованных с положительными результатами варьировала в широких пределах: от 0,20 до 5,25 lg копий/105 клеток (рис. 1).
Только в 1 случае (женщина, 41 год) с высоким уровнем вирусной ДНК в цельной крови (5,25 lg копий/105 клеток) хиВГЧ6-В, передаваемый по наследству, подтвержден анализом волосяных луковиц и ногтевых пластин. Видовая идентификация вируса определена на основании данных, полученных при массовом параллельном секвенировании.
Для расшифровки случая наследственной передачи проведено обследование 3 ближайших родственников женщины с хиВГЧ-6В-статусом. Необходимо подчеркнуть, что на момент лабораторного обследования все пациенты были условно здоровы и жалоб не предъявляли. Отца женщины с хиВГЧ-6В-статусом обследовать не удалось, а у ее супруга проведено только исследование образцов волосяных фолликулов и ногтевых пластин методом ПЦР-РВ. Полученные данные представлены в таблице.
Из таблицы видно, что у 3 из 4 обследуемых членов одной семьи удалось подтвердить наследуемую хромосомную интеграцию вируса. Установление хиВГЧ-6А/В-статуса основано на обнаружении вирусной ДНК в образцах волосяных фолликулов и ногтевых пластин методом ПЦР-РВ. Концентрация ДНК эндогенного вируса у обследуемых с хиВГЧ-6В-статусом (женщины, выявленной ранее в ходе скрининга, ее матери и сына) в зависимости от вида исследуемого биологического материала в среднем составляла: в крови – 5,25 ± 0,04 lg копий/105 клеток, в мазках из ротоглотки – 5,49 ± 0,15; в моче – 5,49 ± 0,16; в волосяных фолликулах – 5,59 ± 0,23, в ногтевых пластинах – 6,03 ± 0,41. Отсутствие у супруга ДНК ВГЧ-6А/В в образцах волосяных фолликулов и ногтевых пластин полностью исключило его хиВГЧ-6А/В-статус, поэтому дальнейшее исследование других видов биологического материала у него было признано нецелесообразным.

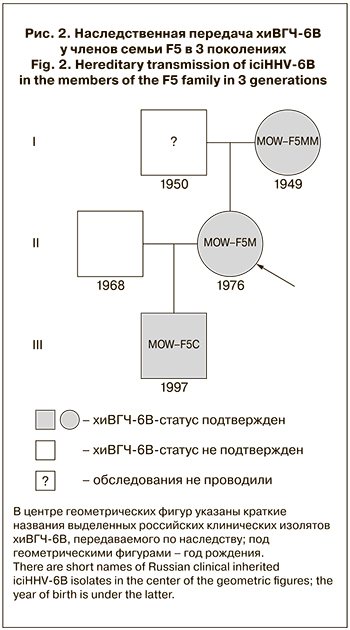
Таким образом, хиВГЧ-6В, передаваемый по наследству, обнаружен и подтвержден результатами ПЦР-анализа волосяных луковиц и ногтевых пластин в 3 поколениях одной семьи: мать–дочь–внук (рис. 2).
 При изучении филогенетического положения геномов 3 российских клинических изолятов хиВГЧ-6В в контексте глобального генетического разнообразия ВГЧ-6В в мире проведен филогенетический анализ на основе сравнения профилей нуклеотидных вариаций, выявленных среди 29 геномов эндогенных штаммов/изолятов (MOW-F5М, MOW-F5ММ, MOW-F5С и 26 ранее депонированных в GenBank NCBI) и 2 эталонных экзогенных штаммов ВГЧ-6В (GenBank NCBI). Общее число позиций, содержащих филогенетически значимые вариации, составило 1240. Филогеномный анализ показал, что российские клинические изоляты MOW-F5М, MOW-F5ММ, MOW-F5С, выделенные в Москве в 2018 г., принадлежали к ВГЧ-6B и образовали уникальную монофилитическую ветвь в пределах клады, в которую вошли преимущественно изоляты хиВГЧ-6В из из Европы (рис. 3).
При изучении филогенетического положения геномов 3 российских клинических изолятов хиВГЧ-6В в контексте глобального генетического разнообразия ВГЧ-6В в мире проведен филогенетический анализ на основе сравнения профилей нуклеотидных вариаций, выявленных среди 29 геномов эндогенных штаммов/изолятов (MOW-F5М, MOW-F5ММ, MOW-F5С и 26 ранее депонированных в GenBank NCBI) и 2 эталонных экзогенных штаммов ВГЧ-6В (GenBank NCBI). Общее число позиций, содержащих филогенетически значимые вариации, составило 1240. Филогеномный анализ показал, что российские клинические изоляты MOW-F5М, MOW-F5ММ, MOW-F5С, выделенные в Москве в 2018 г., принадлежали к ВГЧ-6B и образовали уникальную монофилитическую ветвь в пределах клады, в которую вошли преимущественно изоляты хиВГЧ-6В из из Европы (рис. 3).
В результате опроса женщины с хиВГЧ-6В-статусом установлено, что ее ближайшие родственники по материнской линии (бабушка и дедушка), а также их прародители родом из деревень Бахтино (бабушка) и Смыково (дедушка) Судогодского района Владимирской области. Достоверных официальных данных о состоянии их здоровья и причине смерти получить не удалось.
Обсуждение
Впервые о присутствии полноразмерного интегрированного ВГЧ-6 в ДНК свежеизолированных мононуклеарных клеток периферической крови 3 пациентов сообщили в 1993 г. M. Luppi и соавт. [11]. Далее возможность эффективного встраивания генома вируса в ДНК клетки-хозяина, получившая название хромосомной интеграции, была описана как in vitro, так и in vivo [2, 12, 13].
За более чем 25-летний период изучения наследуемой хромосомной интеграции ВГЧ-6А/В исследования зарубежных авторов были посвящены пониманию не только механизмов встраивания, но и ее последствий для эволюции вирусного генома, теломер человека, а также возможному риску развития патологических состояний и заболеваний, связанных с хиВГЧ-6А/В-статусом [3, 9, 14–17]. Ряд научных работ был направлен на определение частоты выявления носительства эндогенного хиВГЧ-6А/В среди населения в разных странах мира [18–21] и на расшифровку случаев внутрисемейной наследственной передачи от поколения к поколению [20, 22].

Основными критериями при подозрении на хиВГЧ-6А/В-статус, по мнению большинства зарубежных и отечественных ученых, являются обнаружение ДНК ВГЧ-6А/В в крови пациента в концентрации ≥ 5 lg копий/105 клеток или ≥ 5,5–6 lg копий/мл, сохраняющаяся в течение длительного времени, и обнаружение ДНК ВГЧ-6А/В в волосяных фолликулах и ногтевых пластинах при отсутствии клинических проявлений активных форм ВГЧ-6-инфекции [1, 6, 8, 10, 14].
Представленные сведения о распространенности хиВГЧ-6В в Российской Федерации (на примере Москвы и Московской области) в настоящей работе в целом согласуются с мировыми данными, полученными в разных странах мира. Так при тестировании доноров крови в Лондоне (Великобритания) установлена частота выявления наследуемого хиВГЧ-6В, равная 0,8% [18]. В Чешской Республике при обследовании такой же кагорты (здоровых доноров) распространенность хиВГЧ-6А и -В, передаваемых по наследству, составила 0,95% [20], а в США – 0,96% [23]. В 2018 г. H. Miura и соавт. [21] установили распространенность эндогенных хиВГЧ-6А/В в Японии, равную 0,6%. При этом пациентов с хиВГЧ-6А-статусом выявляли чаще, чем с -6В-статусом – в 57 и 43% случаев соответственно [21]. В нашем исследовании частоту выявления хиВГЧ-6А, передаваемого по наследству, установить не удалось, что в большей мере обусловлено недостаточностью количественной репрезентативности выборки.
В целом следует признать, что наследование ВГЧ-6A или ВГЧ-6B, интегрированных в теломеры хромосом человека, происходит с низкой частотой в большинстве популяций, исследованных до настоящего времени, и их характеристики изучены еще недостаточно.
Данные, полученные нами при проведении филогеномного анализа, не противоречат предположению о региональной вариативности геномов хиВГЧ-6В, передаваемых по наследству. Так, Е. Zhang и соавт. [24] показали, что геномы наследуемых хиВГЧ-6В, выделенные в Европе, более тесно связаны друг с другом, чем с геномами эндогенных хиВГЧ-6В из Китая и Пакистана, и значительно отличаются от современных экзогенных штаммов. По мнению авторов, по крайней мере, 1 группа европейских носителей хиВГЧ-6B, включенных в представленный ими анализ, унаследовала один и тот же геном, интегрированный в одну и ту же аллель теломер, от общего предка, предположительно существовавшего 24 500 ± 10 600 лет назад.
Следует подчеркнуть, что на текущий момент в Российской Федерации не накоплено научных данных, демонстрирующих распространенность наследуемых хиВГЧ-6А и хиВГЧ-6В среди населения, описывающих филогенетические особенности циркулирующих клинических изолятов/штаммов хиВГЧ-6А/В; не определены основные сайты интеграции эндогенных вирусов, наиболее часто выявляющиеся на территории нашей страны, что, безусловно, требует более глубокого детального изучения.
Заключение
Впервые получены предварительные данные о частоте выявления хиВГЧ-6A/B, передаваемого по наследству, в Российской Федерации. Распространенность наследуемого хиВГЧ-6B составила 0,4% (1/262; 95% ДИ 0–2,1). Передаваемый по наследству хиВГЧ-6B обнаружен и лабораторно подтвержден в 3 поколениях. Проведенный нами филогеномный анализ показал, что выделенные российские клинические эндогенные изоляты сформировали уникальную монофилитическую ветвь в пределах клады с изолятами хиВГЧ-6В преимущественно из Европы. В ходе исследования эндогенный хиВГЧ-6А не был выявлен. Представленные данные необходимы для понимания генетического разнообразия и географической стратификации хиВГЧ-6A и -6B, передаваемых по наследству, в Российской Федерации и мире.
Нуклеотидная последовательность клинического изолята Human betaherpesvirus 6В MOW-F5C, полученная в ходе настоящей работы, депонирована в базу данных NCBI GenBank (http://www.ncbi.nlm.nih.gov/) под номером MN242397.1.



